
【医学影像 AI】使用深度学习系统检测 9 种婴儿眼底疾病和病症
论文 “使用深度学习系统自动检测视网膜图像中的 9 种婴儿眼底疾病和病症(2024)”,开发了婴儿视网膜智能诊断系统(IRIDS),能够自动检测 9种眼底疾病:早产儿视网膜病变(ROP)(轻度ROP、中度ROP和重度ROP)、视网膜母细胞瘤(RB)、视网膜色素变性(RP)、Coats病、脉络膜缺损、先天性视网膜皱襞
【医学影像 AI】使用深度学习系统检测 9 种婴儿眼底疾病和病症
0. 论文简介
0.1 基本信息
2024年,暨南大学、深圳眼科医院的 Yaling Liu 等在 Nature 子刊 Sci Rep. 发表论文 “使用深度学习系统自动检测视网膜图像中的 9 种婴儿眼底疾病和病症(Automated detection of nine infantile fundus diseases and conditions in retinal images using a deep learning system)”。
本文通过使用来自四家医院的 7697 张视网膜图像(1089名婴儿)的数据集,开发了婴儿视网膜智能诊断系统(Infant Retinal Intelligent Diagnosis System, IRIDS),这是一个自动化系统,能够识别九种眼底疾病和状况,包括早产儿视网膜病变(ROP)(轻度ROP、中度ROP和重度ROP)、视网膜母细胞瘤(RB)、视网膜色素变性(RP)、Coats病、脉络膜缺损、先天性视网膜皱襞(CRF)以及正常眼底。
论文下载: springer
引用格式:
Liu, Y., Xie, H., Zhao, X. et al. Automated detection of nine infantile fundus diseases and conditions in retinal images using a deep learning system. EPMA Journal 15, 39–51 (2024). https://doi.org/10.1007/s13167-024-00350-y

0.2 摘要
目的:
我们开发了婴儿视网膜智能诊断系统(Infant Retinal Intelligent Diagnosis System, IRIDS),旨在帮助早期诊断和监测婴儿眼底疾病及健康状况,以满足眼科医生的迫切需求。
方法:
- 我们通过结合卷积神经网络(CNN)和Transformer结构开发了IRIDS,使用了来自四家医院的 7697 张视网膜图像(1089名婴儿)的数据集。
- 该系统能够识别九种眼底疾病和状况,包括早产儿视网膜病变(ROP, retinopathy of prematurity)(轻度ROP、中度ROP和重度ROP)、视网膜母细胞瘤(RB)、视网膜色素变性(RP)、Coats病、脉络膜缺损、先天性视网膜皱襞(CRF)以及正常眼底。
- IRIDS还包含深度注意力模块、ResNet-18(Res-18)和多轴视觉Transformer(MaxViT)。使用450张视网膜图像将IRIDS的性能与眼科医生进行了比较。IRIDS采用五折交叉验证方法生成分类结果。
结果:
多个基线模型实现了以下指标:准确率、精确率、召回率、F1分数(F1)、Kappa系数和接收者操作特征曲线下面积(AUC),最佳值分别为94.62%(95% CI,94.34%-94.90%)、94.07%(95% CI,93.32%-94.82%)、90.56%(95% CI,88.64%-92.48%)、92.34%(95% CI,91.87%-92.81%)、91.15%(95% CI,90.37%-91.93%)和99.08%(95% CI,99.07%-99.09%)。相比之下,IRIDS在测试数据集上的多标签分类中表现优异,使用Res-18和MaxViT模型的平均准确率、精确率、召回率、F1分数、Kappa系数和AUC分别为96.45%(95% CI,96.37%-96.53%)、95.86%(95% CI,94.56%-97.16%)、94.37%(95% CI,93.95%-94.79%)、95.03%(95% CI,94.45%-95.61%)、94.43%(95% CI,93.96%-94.90%)和99.51%(95% CI,99.51%-99.51%)。这些结果表明,特别是在AUC方面,IRIDS的表现值得进一步研究其在视网膜异常检测中的应用。
结论:
IRIDS能够准确识别九种婴儿眼底疾病和状况。它可以帮助医疗资源不足地区的非眼科专业人员进行婴儿眼底疾病筛查,从而预防严重并发症。IRIDS是人工智能与眼科结合的典范,有助于在婴儿眼底疾病的治疗中实现更好的预测性、预防性和个性化医学(PPPM / 3PM)效果。
1. 引言
婴儿眼底疾病的早期诊断和治疗是提高患儿及其家庭生活质量的关键因素。常见的婴儿眼底疾病包括早产儿视网膜病变(ROP)[1]、Coats病[2, 3]、视网膜母细胞瘤(RB)[4]、色素性视网膜炎(RP)[5]、脉络膜缺损[6]、先天性视网膜畸形(CRF)[7]以及家族性渗出性玻璃体视网膜病变[8]。
如果这些疾病的诊断和管理被延迟,可能会降低治疗效果,并导致严重的甚至不可逆的结构和功能并发症,例如屈光不正、夜盲症、斜视和新生血管青光眼[2, 9]。识别高危个体是预防这些严重并发症的关键步骤,这一点在欧洲协会关于PPPM(即产前-围生期-产后医疗)的白皮书中得到了强调[10]。
早产儿视网膜病变(ROP, retinopathy of prematurity)是婴儿眼底异常的典型例子,是早年视觉损伤和失明的主要原因之一,需要及时诊断和干预[11]。全球每年约有8000例婴儿RB病例需要进行眼球摘除手术[12–14]。由于某些婴儿眼底疾病较为罕见,在一些儿童医院和妇幼保健医院中,即使遇到这些病例,做出准确诊断也可能具有挑战性。这不仅对个人及其家庭产生负面影响,还可能对整个社会造成危害。然而,全球许多资源匮乏的地区缺乏经验丰富的眼科医生[15],使得患有眼底疾病的婴儿面临立即的风险。因此,开发一种独特的策略,遵循PPPM原则,以实现婴儿眼底疾病早期检测变得至关重要[16]。
因此,可以利用最新的数字计算技术创新来开发有效的自动化诊断工具,以便对婴儿眼底疾病进行及时的诊断和管理。深度学习(DL)是一项成熟且迅速发展的技术,尤其是在人类疾病的计算机辅助诊断领域[17–19]。这一前沿领域正在扩展到医学的其他领域,例如临床实践、转化医学研究和基础生物医学研究[20–24]。深度学习在自动化筛查和诊断各种眼底疾病方面表现出色,包括糖尿病视网膜病变、年龄相关性黄斑变性、屈光不正、青光眼和视网膜肿瘤等。其中一些应用正在逐步向临床实施推进[25–29]。
关于婴儿眼底疾病,我们团队成功地使用深度学习来识别ROP、ROP的I区以及A-ROP,并有效地应用了增强算法来进行视网膜分级[30–34]。大多数相关研究都集中在利用视网膜图像检测单一的眼底疾病上。最近,研究人员开发了基于深度学习辅助系统来检测多种眼底疾病[35, 36],其中一种系统使用智能手机的广角视网膜成像系统对儿童进行多病种分类[37]。值得注意的是,上述研究主要集中在单一眼底疾病的检测上,或者侧重于成人和较大儿童的眼底图像分析。
研究的假设与目的
迄今为止,尚未有涉及多发病变检测的深度学习研究开展。在临床实践中,尤其是在缺乏专业眼科医生的偏远地区,这样的婴儿眼底疾病多病检测系统不仅必要,而且可能非常有益。许多婴儿眼底疾病难以被家长察觉,通常是在孩子因弱视等其他问题就诊时才被发现。此外,婴儿眼底疾病的诊断具有挑战性,导致严重并发症的发生率增加。
我们假设通过利用深度学习模型,自动分析视网膜图像来检测婴儿眼底疾病,可以提高对患儿的诊断率,预防严重并发症的发生,并最终提升其生活质量。我们认为这种方法可以在临床中得到有效应用,为筛查和管理婴儿眼底疾病提供更准确、可靠的工具。
为了实现这一目标,我们开发了一个名为“婴儿视网膜智能诊断系统”(Infantile Retinal Intelligence Diagnosis System, IRIDS)的自动化疾病检测系统。该系统能够利用来自中国各地医院收集到的7679张视网膜图像,对九种常见的婴儿眼底疾病和状况进行分类。
我们期待这一系统能够在预防可避免的婴儿眼底疾病方面带来显著的个人结果改善,并对人群高级医疗服务产生积极的成本效益影响。这包括采用创新的深度学习筛选技术,结合针对每位患者个性化配置文件的预测性疾病模型和治疗算法[38]。
2. 方法
2.1 数据集与标注
为了开发和验证婴儿视网膜智能诊断系统(IRIDS),我们从四个不同的数据源收集了视网膜图像。
用于训练、验证和测试的主要数据集来自中国广东省深圳市眼科医院眼科部,采集时间为2004年4月至2021年1月。一小部分数据来自广东省的三家其他妇幼保健医院(揭阳市妇幼保健院、桂林市妇幼保健院、广东省妇幼保健院)。这项诊断研究得到了深圳眼科医院伦理委员会的批准。所有机构都遵循了《赫尔辛基宣言》的原则[39],并从参与婴儿的所有父母那里获得了书面知情同意。
数据采集和处理方法见图1A和补充图2。
来自不同数据源的所有图像均经过两位年轻眼科医生的质量筛选。为了在不同类别之间保持平衡,并非所有图像都被纳入数据库。例如,如果ROP(视网膜病变)的图像数量过多,则只选择一部分具有代表性的图像,以确保数据多样性,同时仍保证总图像数量足够。随后,质量不佳的图像(如模糊、过暗或过亮)被排除在数据库之外。总共在标注前排除了 2933 张图像。
纳入的图像由两位经过培训的年轻眼科医生独立进行标注,以确保 IRIDS 算法分类的准确性。对于标签存在冲突的图像,需提交资深眼科医生复审,并根据经验丰富的视网膜专科医生的意见确定最终标签。最后,具有统一标签的图像被保留下来,用于自动系统的训练、验证和测试数据集。
由于某些图像无法分类(特征不确定)或属于不在九个类别中的罕见疾病,这些图像也被排除在分析之外。这导致额外排除了1874 张图像,最终纳入的数据集包含 7697 张图像。


2.2 类别
所有视网膜图像根据《儿科视网膜》[40] 中描述的视网膜图像特征被分类为九种主要疾病和状况(补充表1 和补充图1)。具有独特视网膜特征的常见视网膜疾病,这些特征在视网膜图像上可以识别,被归类为独立的主要类别。为了反映实际情况,我们还包括了正常视网膜图像。
这些主要类别包括 :ROP(早产儿视网膜病变)、RP(色素变性)、RB(视网膜母细胞瘤)、Coats 病(外层渗出性视网膜病变)、脉络膜缺损(choroid coloboma)、先天性视网膜皱褶(Congenital Retinal Folds)和 正常(Normal)的视网膜图像。

一些疾病具有相似的特征,仅通过视网膜图像很难区分。例如,患有 1 或 2 期 ROP 的婴儿症状较轻,而其他阶段的患儿症状则更为严重。为此,我们将它们归为一类,并将其分类为“轻度”ROP。同样地,患有 3 期 ROP 的婴儿更可能需要激光光凝术或抗血管内皮生长因子玻璃体内注射治疗,而较轻的 ROP 则不需要这些治疗;因此我们将这一阶段归类为“中度”ROP。4 和 5 期 ROP 的患儿通常需要进行玻璃体切除术或其他手术治疗,因此我们将它们归为一类,并分类为“重度”ROP。
经过这种聚类后,九种疾病和状况被划分到七个主要类别和三个子类别中。每个类别都被赋予了唯一的类别识别号(ID)(补充表1)。

2.3 IRIDS 的系统架构
图 1B 和 1C 展示了 IRIDS 的工作流程和系统架构。
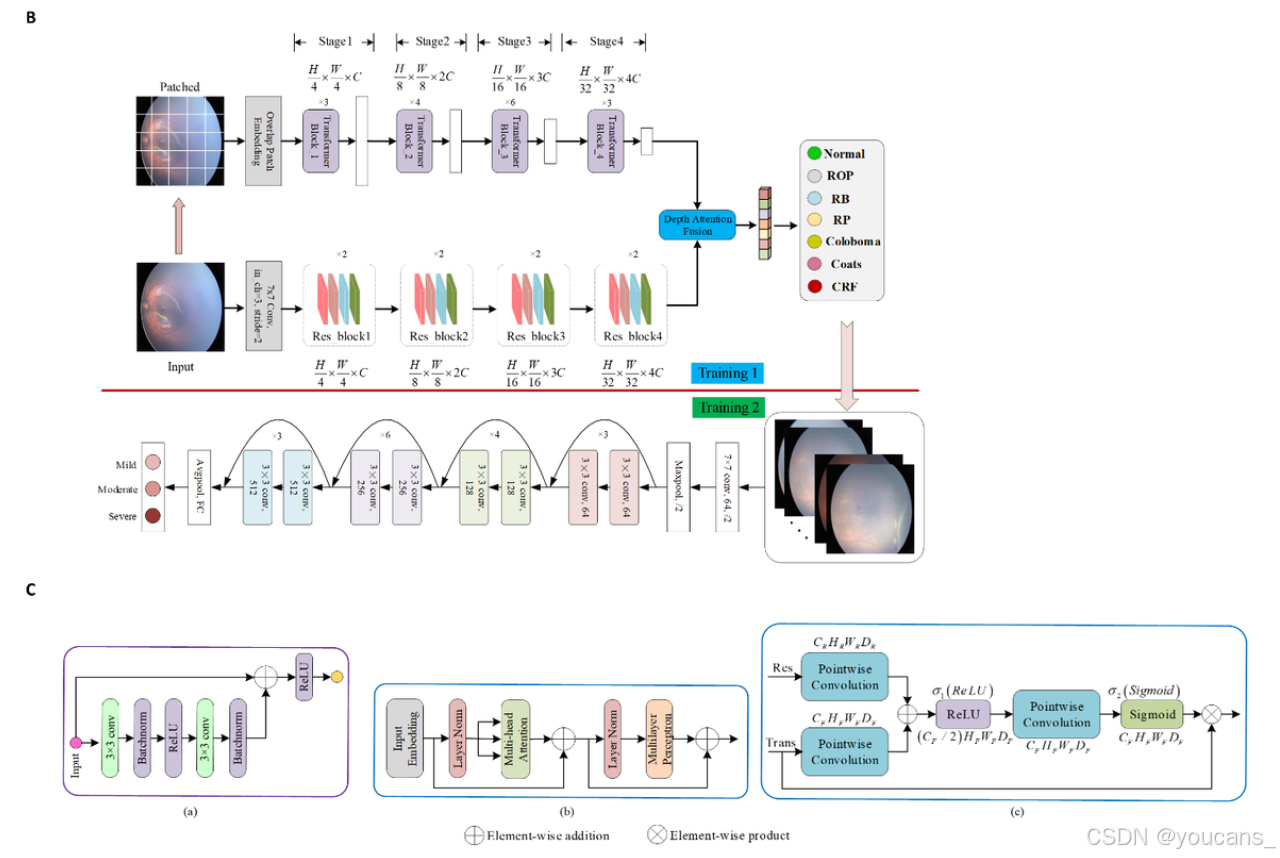
图1:研究流程和 IRIDS 架构
B. IRIDS 架构
IRIDS 包含两个分支:卷积神经网络(CNNs)和变压器网络(Transformers)。该模型采用四阶段变压器模块提取全局特征,并选择了带残差块的 ResNet-34 来提取局部特征。提取到的特征通过深度注意力融合模块进行融合,以充分表示与不同疾病相关的特定特征。
C. 使用的模块示意图
(a)、(b) 和 © 分别代表了残差块模块、变压器块和深度注意力融合模块。
卷积神经网络(CNNs)主要关注局部特征,因此对全局特征的表示不够充分,而 Transformers 网络更注重对全局特征的提取。当仅使用一种结构从分析图像中提取特征时,分类性能受到限制。因此,我们采用了一种混合模型,结合了 CNNs 和变压器结构,如图 1B 所示。
ResBlocks 来自 ResNet-18 模型 [41],表示为一个残差块。残差块的定义如图 1C (a) 所示。使用的 Transformers 块与 MaxViT [42] 的相同,其主要 Transformers 结构如图 1C (b) 所示。从 Res-blocks 和 Transformer 块中获得高级特征后,我们使用深度注意力融合模块将来自 ResNet 和 MaxViT 模块路径的局部和全局特征进行融合,如图 1C © 所示。因此,整个网络可以表达与每种特定疾病相关的特征,并专注于提取每种情况的关键特征。
在所有模型中,我们使用 Pytorch 库对数据集进行训练和测试。使用 NVIDIA TITAN XP GPU 加速训练和测试过程。选择 Adam 优化器(betas = (0.9, 0.997),eps = 10−8)作为学生网络的优化器。判别器使用随机梯度下降(SGD)作为优化器,动量为 0.9,权重衰减为 5×10−5。学生和判别器网络的初始学习率均设置为 0.001,然后每 20 个 epoch 乘以 0.1。最大 epoch 设置为 100,小批量的大小设置为 16。所有图像均调整为 224×224 像素,并用作所有网络的输入。
2.4 与眼科医生的性能对比
为了将 IRIDS 的性能与具有临床经验的眼科医生进行比较,我们邀请了两位在视网膜疾病方面拥有 10 到 15 年临床经验的专家和两位有 5 到 10 年经验的资深眼科医生,让他们独立对读者研究中相同的图像进行分类。
为了使比较更加全面和具有代表性,我们使用了一个额外的数据集,该数据集包含 2022 年从深圳眼科医院收集的 450 张视网膜图像。这个数据集不仅涵盖了大类类别(main class),还包含了次级类别(subclass),每个类别都有 50 张图像。这种分类方式能够更细致地评估 IRIDS 的表现。
2.5 统计分析
我们使用多种评估指标来衡量 IRIDS 模型的性能,包括准确率、精确率、召回率、kappa 值、受试者工作特征曲线下的面积(AUC)和 F1 分数。
我们使用了多种评估指标来衡量 IRIDS 模型的性能,包括:
- 准确率 (Accuracy):模型预测正确的样本占总样本的比例。
- 精确率 (Precision):模型预测为正类的样本中,实际为正类的比例。
- 召回率 (Recall):实际为正类的样本中,被模型正确预测为正类的比例。
- kappa 值:用于评估分类模型的可靠性,考虑了随机一致性的影响。
- AUC(Area Under the ROC Curve):ROC 曲线下的面积,用于评估模型在不同阈值下的分类性能,ROC 越接近 1 则模型性能越好。
- F1 分数 (F1-score):精确率和召回率的调和平均数,用于平衡两者之间的关系。
我们采用了五折交叉验证(5-fold cross-validation)方法,以获取所有类别上的分类结果,并对每折(fold)的结果进行计算和分析,以评估模型的整体性能。
说明:这种方法(5-fold cross-validation)将数据集分为5个子集,依次使用其中4个子集作为训练集,剩下的1个子集作为测试集,重复5次,确保每个子集都被用作测试集。这种方法可以有效利用数据,减少模型评估的偏差和方差。
3. 结果
3.1 数据特征与预测系统架构
整个工作流程、IRIDS的简化架构以及主要数据集处理流程如图1A、1B和补充图2所示。
总共收集了7697张图像(1089个病例),用于模型训练(80%)和测试(20%)。图像数据的分布以及每个数据集各类别的结果如图1A、补充图2和补充表2所示。

3.2 数据测试中的性能
首先,我们使用基线模型进行了多项对比实验。分类结果列于表1中。
在CNN模型中,Res-18模 型取得了最佳的分类性能(表1)。在Transformer模型中,MaxViT模型在所有分类指标中均取得了最高的准确率和精确率。因此,选择Res-18模型提取局部上下文特征信息,并考虑到其低复杂性和高分类性能,采用MaxViT模型提取全局特征。
此外,我们绘制了这些基线方法的受试者工作特征(ROC)曲线以比较模型性能。从中发现,Res-18和MaxViT模型表现最佳(图2A)。

3.3 高级测试中的性能
通过基线方法的对比实验,选择Res-18和MaxViT模型作为构建IRIDS的主要结构。
为了融合从Res模块和Transformer模块中提取的特征,我们采用了一种深度注意力(DA)融合模块来高效融合高级特征。因此,IRIDS由Res-18、MaxViT和DA模块组成。
我们对这些模块进行了消融实验以验证其有效性;Res-18 + MaxViT + DA代表所提出的方法。实验结果如表2 所示。我们提出的方法取得了最佳的分类性能,准确率、精确率、召回率、F1分数、Kappa系数和AUC分别为96.45%、95.86%、94.37%、95.03%、94.43%和99.51%。
我们观察到,IRIDS 在所有评估指标中均取得了最佳的ROC性能和最高的AUC(图2B 和3A)。特别是对于 RP 和 CRF,所提出方法的正确预测概率比其他对比方法至少高出五个百分点。使用该方法,正常类别的所有图像均被正确检测。

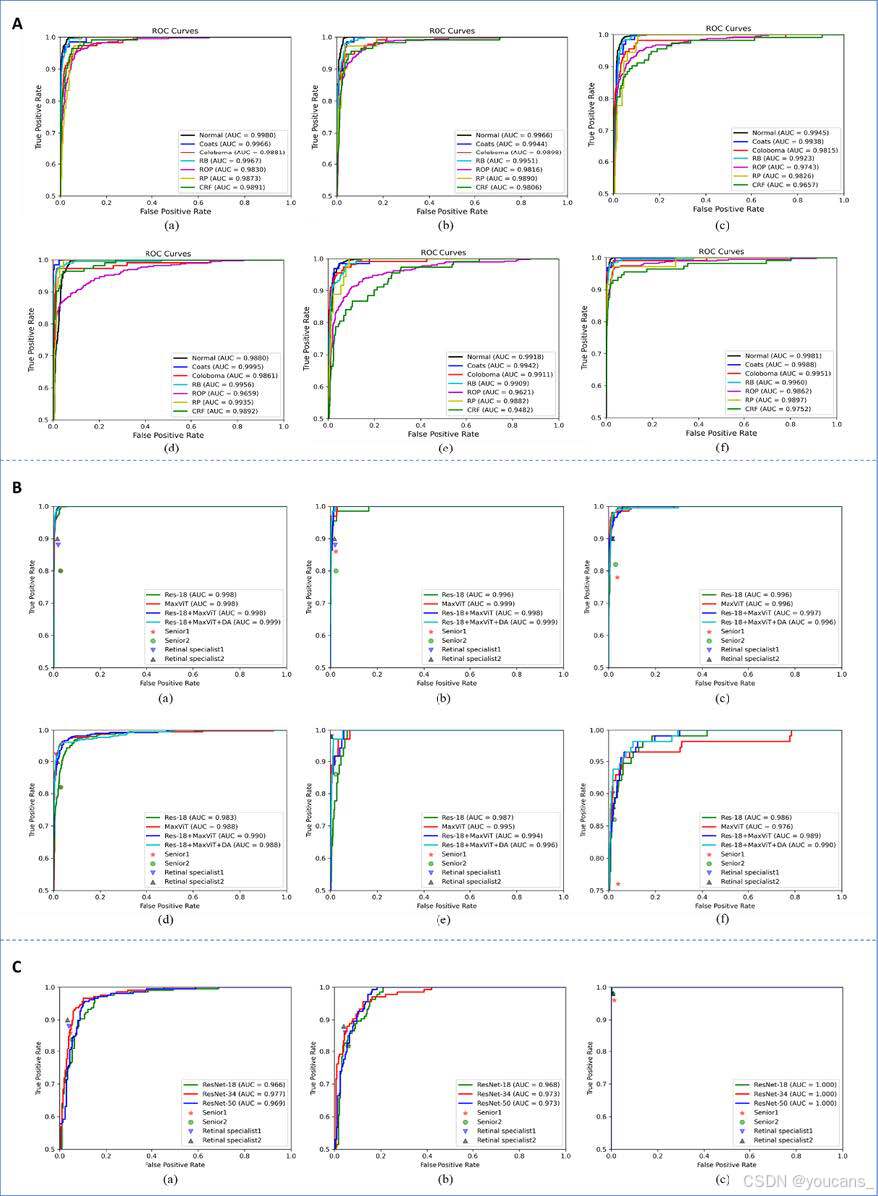
图2:本研究分析的每种深度学习方法的ROC曲线。
A.不同基线方法的ROC曲线。(a)-(f) 分别表示Res-18、Res-34、Res-50、ViT small、RegionViT和MaxViT模型。
B.主要疾病和病症类别的比较方法的ROC曲线。(a)-(f) 分别代表Coats、Coloboma、RB、ROP、RP和CRF类别。
C.ROP分期比较方法的ROC曲线。(a)-© 分别代表轻度、中度和重度条件。

图3:分类器性能的可视化。
A.不同方法的混淆矩阵。(a)-(d) 分别表示 Res-18、MaxViT、Res-18+MaxViT 和 Res-18+Max ViT+DA(提出的)模型。
B.不同方法的T-SNE可视化。(a)-(d) 分别表示 Res-18、MaxViT、Res-18+MaxViT 和 Res-18+Max ViT+DA(提出的)模型。
3.4 T 分布随机邻域嵌入(T-SNE)可视化
我们通过将最后特征提取层的特征应用于所有对比方法,进行了T-SNE可视化,以证明所提出方法提取特征的有效性,如图3B所示。我们观察到,与其他方法相比,所提出的方法能够更好地分离所有预测类别(图3B)。
3.5 IRIDS与眼科医生的比较
IRIDS与眼科医生的比较结果如图2B所示。
对于六种主要眼底疾病的诊断,IRIDS的AUC值范围为0.976至0.999,其中对于三种疾病(Coats病、Coloboma和RB),IRIDS的AUC值高于或等于视网膜专家的水平。对于其他眼底疾病(RP、ROP和CRF),IRIDS的AUC值与视网膜专家相当。与高级眼科医生相比,IRIDS在六种主要疾病类别中均表现出更高的AUC值。
此外,我们对ROP分期模型进行了人机比较(图2C和补充表3)。结果显示,对于严重ROP,IRIDS的表现优于眼科医生,但对于其他ROP类别,差异不显著。
4. 讨论
**在本研究中,我们开发了IRIDS模型,用于自动识别九种婴儿眼底疾病和病变。通过对比多种算法,我们确定该模型具有最高的性能,AUC值达到99.51%。**此外,五折交叉验证进一步证实了模型的优异性能。与视网膜专家相比,IRIDS表现出更高的诊断准确性。
我们首先使用基线模型进行了多项对比实验,其结果如表1所示。最终,我们选择了结合DA模块的Res-18和MaxViT模型作为构建IRIDS的主要结构。IRIDS用于分析从四家医院收集的数据。尽管这些医院的图像质量、成像技术和设备类型可能存在差异,但模型始终表现优异,表明其具有较强的适应性。
许多基于CNN的深度学习方法专注于局部特征提取,而忽略了全视网膜视野中的有价值信息。为了解决这一问题,我们采用了Transformer模型用于视觉任务[45–47],该模型可以从输入图像中提取长距离依赖特征。因此,将CNN和Transformer结构整合到一个模型中,能够同时提取局部和全局特征,从而提升模型的分类性能。
CNN在图像分析中的不透明性可能阻碍临床医生采用深度学习技术[48]。然而,我们的模型能够有效提取与特定疾病相关的关键局部和全局特征,这一点通过T-SNE分析结果得到了验证。这一特性不仅提高了分类性能,还为其在临床实践中的应用提供了潜力。
在本研究中,我们探索了使用IRIDS自动检测婴儿眼底疾病的方法。我们之前曾报道过使用超广角视网膜图像诊断ROP的研究[31]。此外,也有研究针对ROP分期、RB和RP的诊断进行了探索[49–51]。然而,这些研究主要集中在使用视网膜图像检测单一婴儿眼底疾病。随后,越来越多的研究开始转向自动化诊断多种眼底疾病。一些研究尝试使用单一算法区分多种眼底疾病[35]。Stevenson等人[52]开发了一种多类别深度学习模型,利用公开的视网膜图像数据库检测眼底疾病。该研究表明,即使在没有编程或人工智能知识的情况下,使用标准笔记本电脑开发AI分类器也是可行的。Son等人[53]使用更大的数据集构建了检测12种视网膜异常的深度学习模型。其外部测试集的AUC高达98%,表现出卓越的性能。此外,模型的可解释性和可靠的分类输出表明其有潜力作为视网膜图像的自动筛查系统用于临床。由于这些系统检测的是异常而非特定眼底疾病,因此这些模型不能直接用于筛查。Lin等人[54]开发了一种基于深度学习的系统,前瞻性评估了18,163张彩色眼底照片,检测了14种眼底疾病,AUC达到0.98。Li等人[35]开发并前瞻性验证了一种基于视网膜图像的深度学习算法,可同时识别临床实践中的十种眼底疾病。这与通用筛查方法一致,具有实用性。Cen等人[55]使用包含60万张视网膜图像的大型数据库,建立了一个数字光处理系统,对39种眼底疾病和病变的检测具有较高的F1分数、敏感性、特异性和AUC。Ju等人[56]提出了一种新方法,使深度神经网络能够从包含超过一百万张图像的综合性眼底数据库中学习,从而促进多种眼底疾病的识别。Gu等人[57]评估了AI系统在真实世界场景(特别是初级医疗环境)中检测多种眼底疾病的性能,并取得了高准确率。然而,这些研究仅关注成人眼底疾病,未涵盖婴儿眼底疾病。IRIDS利用非侵入性视网膜图像(易于获取)来实现多种婴儿眼底疾病和病变的自动筛查,使其比现有方法更具临床适用性和适应性。
5. 结论、专家建议及3PM框架下的展望
5.1 结论
IRIDS通过利用前沿的深度学习能力,能够准确检测九种婴儿眼底疾病和病变。得益于其卓越的诊断精度和可靠性,IRIDS可以改善全球儿童及其家庭的视觉健康和生活质量,特别是在视网膜专家短缺的地区。它能够通过及时检测视网膜病变,有效识别和预防严重并发症的发生。IRIDS的部署在增强早期检测和干预方面具有巨大潜力,最终有助于改善预后并为有需要的儿童提供更好的医疗服务。
5.2 预测性方法
通过使用深度注意力融合模块融合ResNet和MaxViT模块路径中的局部和全局特征,IRIDS不仅实现了多种婴儿眼底病变和疾病的自动筛查,还展示了与眼科医生(包括视网膜专家)相当甚至更高的诊断准确性。
如补充表1所示,IRIDS能够自动检测婴儿中最常见的眼底疾病和病变,如Coats病、RB、RP、脉络膜缺损和CRF,其AUC值接近1.0,表明其性能优异。
尽管数据不平衡率较高(如多标签设置研究[58]所述),IRIDS的高AUC结果进一步证实了我们模型的高准确性,这可能归因于严格的数据集选择过程[53]。在PPPM(预测性、预防性和个性化医学)框架下[38],将这种新方法纳入当前临床工作流程具有优势,因为它有可能为多种婴儿疾病的诊断提供更加个性化和可量化的方法。这进一步证实了我们模型的鲁棒性和可靠性,使其成为婴儿眼底疾病早期检测和治疗的有力工具。
5.3 针对性预防
在预防的背景下[38],由于IRIDS在人机对比中的出色表现,该系统使我们能够识别并及时治疗一些容易被忽视或误诊的婴儿眼底疾病,即使在没有视网膜专家的情况下也是如此。这有助于防止患者病情的进一步恶化并阻止疾病的进展。IRIDS有助于减轻眼科医生的负担并提高筛查效率,特别是在欠发达地区。
5.4 医疗服务的个性化
我们纳入了来自四家医院的九种不同婴儿眼底疾病和病变。
为了解决使用单一结构从图像分析中提取特征的局限性,我们采用了结合CNN和Transformer结构的混合模型。这种混合模型能够同时关注局部和全局特征。因此,九种疾病分类的AUC(曲线下面积)达到了令人印象深刻的99.51%。最终,整个网络能够捕捉疾病特异性特征并专注于提取每种病变的关键特征。因此,IRIDS可以基于单张视网膜图像对个体婴儿进行诊断。
由于其简单性和易于训练的特点,IRIDS还可以在没有专业眼科医生的环境中应用,有助于解决诊断不足、误诊和过度检查的问题。
5.5 PPPM框架下的局限性与展望
本研究存在一些局限性。
首先,与许多其他眼底疾病类似,一些婴儿眼底疾病缺乏通用的诊断标准或共识。缓解这一问题的方法可以包括参考多个权威来源或相关研究。
其次,尽管我们收集了多达九种常见婴儿眼底疾病和病变的数据,但并未涵盖所有婴儿眼底疾病或病变。由于图像数据不足,家族性渗出性玻璃体视网膜病变和视网膜脱离等疾病未被纳入。添加“其他”类别可能是解决这一局限性的有效方法。
第三,我们的研究数据集相对较小且仅限于单一地理区域。此外,该数据集仅包含中国个体,限制了其普适性。因此,应纳入来自世界其他地区数据库的图像,以增加多样性并扩展数据库的适用性。
该深度学习系统是首个以非侵入性方式自动识别婴儿眼底疾病的系统。它还符合2019年EPMA世界大会强调的PPPM方法的关键方面,即通过定制和持续监测患者的临床参数来改善治疗结果[59]。
为了促进深度学习在该领域的未来应用,强烈建议考虑以下几点:
- 确保全面检测多种类别的婴儿眼底疾病或病变,同时实现适用于每个特定类别的智能临床诊断。
- 实现疾病进展的全面预测,并促进与临床环境相关的治疗决策评估,从而在临床监测中早期应用。通过PPPM方法,旨在从诊断到治疗决策提供完整的预测,从而为患者提供更好的临床结果。
总结来说,IRIDS作为一种创新的深度学习系统,在婴儿眼底疾病的自动检测和预防性医疗中展现了巨大潜力。通过进一步优化和扩展,它有望成为临床实践中的重要工具,为患者提供更精准和个性化的医疗服务。
6. 数据
本诊断研究已获得深圳眼科医院伦理委员会的批准。所有机构都遵守《赫尔辛基宣言》的宗旨。所有入选婴儿的父母均已签署书面知情同意书。同意发表本文已获作者批准发表。
7. 参考文献
- Chiang MF, Quinn GE, Fielder AR, Ostmo SR, Paul Chan RV, Berrocal A, et al. International Classification of Retinopathy of Prematurity, Third Edition. Ophthalmology. 2021;128(10):e51–68. https://doi.org/10.1016/j.ophtha.2021.05.031.
- Shields JA, Shields CL, Honavar SG, Demirci H. Clinical variations and complications of Coats disease in 150 cases: the 2000 Sanford Gifford Memorial Lecture. Am J Ophthalmol. 2001;131(5):561–71. https://doi.org/10.1016/s0002-9394(00)00883-7.
- Spitznas M, Joussen F, Wessing A, Meyer-Schwickerath G. Coat’s disease. An epidemiologic and Fluorescein angiographic study. Albrecht Von Graefes Arch Klin Exp Ophthalmol. 1975;195(4):241–50. https://doi.org/10.1007/BF00414937.
- Rao R, Honavar SG. Retinoblastoma. Indian J Pediatr. 2017;84(12):937–44. https://doi.org/10.1007/s12098-017-2395-0.
- Pagon RA. Retinitis pigmentosa. Surv Ophthalmol. 1988;33(3):137–77. https://doi.org/10.1016/0039-6257(88)90085-9.
- Giles K, Raoul C, Yannick B, Peter W. Uveal coloboma: about 3 cases at the University Teaching Hospital, Yaounde, Cameroon. Pan Afr Med J. 2016;24:201. https://doi.org/10.11604/pamj.2016.24.201.9770.
- Nishina S, Suzuki Y, Yokoi T, Kobayashi Y, Noda E, Azuma N. Clinical features of congenital retinal folds. Am J Ophthalmol. 2012;153(1):81-7 e1. https://doi.org/10.1016/j.ajo.2011.06.002.
- Liche F, Majji AB. Familial exudative vitreoretinopathy. Ophthalmology. 2012;119(5):1093. https://doi.org/10.1016/j.ophtha.2012.02.025.
- Fielder A, Blencowe H, O’Connor A, Gilbert C. Impact of retinopathy of prematurity on ocular structures and visual functions. Arch Dis Child Fetal Neonatal Ed. 2015;100(2):F179–84. https://doi.org/10.1136/archdischild-2014-306207.
- Golubnitschaja O, Costigliola V, Epma. General report & recommendations in predictive, preventive and personalised medicine 2012: white paper of the European Association for Predictive, Preventive and Personalised Medicine. EPMA J. 2012;3(1):14. https://doi.org/10.1186/1878-5085-3-14.
- Good WV. Retinopathy of Prematurity Incidence in Children. Ophthalmology. 2020;127(4S):S82–3. https://doi.org/10.1016/j.ophtha.2019.11.026.
- Dimaras H, Corson TW, Cobrinik D, White A, Zhao J, Munier FL, et al. Retinoblastoma. Nat Rev Dis Primers. 2015;1:15021. https://doi.org/10.1038/nrdp.2015.21.
- Global Retinoblastoma Study G, Fabian ID, Abdallah E, Abdullahi SU, Abdulqader RA, Adamou Boubacar S, et al. Global Retinoblastoma Presentation and Analysis by National Income Level. JAMA Oncol. 2020;6(5):685–95. https://doi.org/10.1001/jamaoncol.2019.6716.
- Chen HY, Lehmann OJ, Swaroop A. Genetics and therapy for pediatric eye diseases. EBioMedicine. 2021;67:103360. https://doi.org/10.1016/j.ebiom.2021.103360.
- Coleman K, Coleman J, Franco-Penya H, Hamroush F, Murtagh P, Fitzpatrick P, et al. A New Smartphone-Based Optic Nerve Head Biometric for Verification and Change Detection. Transl Vis Sci Technol. 2021;10(8):1. https://doi.org/10.1167/tvst.10.8.1.
- Golubnitschaja O, Potuznik P, Polivka J Jr, Pesta M, Kaverina O, Pieper CC, et al. Ischemic stroke of unclear aetiology: a case-by-case analysis and call for a multi-professional predictive, preventive and personalised approach. EPMA J. 2022;13(4):535–45. https://doi.org/10.1007/s13167-022-00307-z.
- Topol EJ. High-performance medicine: the convergence of human and artificial intelligence. Nat Med. 2019;25(1):44–56. https://doi.org/10.1038/s41591-018-0300-7.
- Baek SU, Lee WJ, Park KH, Choi HJ. Health screening program revealed risk factors associated with development and progression of papillomacular bundle defect. EPMA J. 2021;12(1):41–55. https://doi.org/10.1007/s13167-021-00235-4.
- Li S, Li M, Wu J, Li Y, Han J, Cao W, et al. Development and validation of a routine blood parameters-based model for screening the occurrence of retinal detachment in high myopia in the context of PPPM. EPMA J. 2023;14(2):219–33. https://doi.org/10.1007/s13167-023-00319-3.
- Golub TR, Slonim DK, Tamayo P, Huard C, Gaasenbeek M, Mesirov JP, et al. Molecular classification of cancer: class discovery and class prediction by gene expression monitoring. Science. 1999;286(5439):531–7. https://doi.org/10.1126/science.286.5439.531.
- Wang Y, Tetko IV, Hall MA, Frank E, Facius A, Mayer KF, et al. Gene selection from microarray data for cancer classification–a machine learning approach. Comput Biol Chem. 2005;29(1):37–46. https://doi.org/10.1016/j.compbiolchem.2004.11.001.
- Yu KH, Levine DA, Zhang H, Chan DW, Zhang Z, Snyder M. Predicting Ovarian Cancer Patients’ Clinical Response to Platinum-Based Chemotherapy by Their Tumor Proteomic Signatures. J Proteome Res. 2016;15(8):2455–65. https://doi.org/10.1021/acs.jproteome.5b01129.
- Yu KH, Fitzpatrick MR, Pappas L, Chan W, Kung J, Snyder M. Omics AnalySIs System for PRecision Oncology (OASISPRO): a web-based omics analysis tool for clinical phenotype prediction. Bioinformatics. 2018;34(2):319–20. https://doi.org/10.1093/bioinformatics/btx572.
- Check Hayden E. The automated lab. Nature. 2014;516(7529):131–2. https://doi.org/10.1038/516131a.
- Chew EY. Age-related Macular Degeneration: Nutrition, Genes and Deep Learning-The LXXVI Edward Jackson Memorial Lecture. Am J Ophthalmol. 2020;217:335–47. https://doi.org/10.1016/j.ajo.2020.05.042.
- Shon K, Sung KR, Shin JW. Can Artificial Intelligence Predict Glaucomatous Visual Field Progression? A Spatial-Ordinal Convolutional Neural Network Model. Am J Ophthalmol. 2022;233:124–34. https://doi.org/10.1016/j.ajo.2021.06.025.
- Ee CL, Samsudin A. Comparison of Smartphone-Based and Automated Refraction with Subjective Refraction for Screening of Refractive Errors. Ophthalmic Epidemiol. 2022;29(5):588–94. https://doi.org/10.1080/09286586.2021.1986550.
- Dai L, Wu L, Li H, Cai C, Wu Q, Kong H, et al. A deep learning system for detecting diabetic retinopathy across the disease spectrum. Nat Commun. 2021;12(1):3242. https://doi.org/10.1038/s41467-021-23458-5.
- Panwar N, Huang P, Lee J, Keane PA, Chuan TS, Richhariya A, et al. Fundus Photography in the 21st Century–A Review of Recent Technological Advances and Their Implications for Worldwide Healthcare. Telemed J E Health. 2016;22(3):198–208. https://doi.org/10.1089/tmj.2015.0068.
- Zhao J, Lei B, Wu Z, Zhang Y, Li Y, Wang L, et al. A Deep Learning Framework for Identifying Zone I in RetCam Images. IEEE Access. 2019;7:103530–7. https://doi.org/10.1109/access.2019.2930120.
- Zhang Y, Wang L, Wu Z, Zeng J, Chen Y, Tian R, et al. Development of an Automated Screening System for Retinopathy of Prematurity Using a Deep Neural Network for Wide-Angle Retinal Images. IEEE Access. 2019;7:10232–41. https://doi.org/10.1109/access.2018.2881042.
- Rugang Zhang JZ. Hai Xie, Tianfu Wang, Automatic diagnosis for aggressive posterior retinopathy of prematurity via deep attentive convolutional neural network. Expert Syst Appl. 2022;187:115843.
- Maji D, Sekh AA. Automatic grading of retinal blood vessel in deep retinal image diagnosis. J Med Syst. 2020;44(180). https://doi.org/10.1007/s10916-020-01635-1.
- Xie HLH, Zeng X, He Y, Chen G. AMD-GAN: attention encoder and multi-branch structure based generative adversarial networks for fundus disease detection from scanning laser ophthalmoscopy images. Neural Netw. 2020;132:477–90.
- Dong L, He W, Zhang R, Ge Z, Wang YX, Zhou J, et al. Artificial Intelligence for Screening of Multiple Retinal and Optic Nerve Diseases. JAMA Netw Open. 2022;5(5):e229960. https://doi.org/10.1001/jamanetworkopen.2022.9960.
- Cen LP, Ji J, Lin JW, Ju ST, Lin HJ, Li TP, et al. Automatic detection of 39 fundus diseases and conditions in retinal photographs using deep neural networks. Nat Commun. 2021;12(1):4828. https://doi.org/10.1038/s41467-021-25138-w.
- Patel TP, Kim TN, Yu G, Dedania VS, Lieu P, Qian CX, et al. Smartphone-Based, Rapid, Wide-Field Fundus Photography for Diagnosis of Pediatric Retinal Diseases. Transl Vis Sci Technol. 2019;8(3):29. https://doi.org/10.1167/tvst.8.3.29.
- Golubnitschaja O, Baban B, Boniolo G, Wang W, Bubnov R, Kapalla M, et al. Medicine in the early twenty-first century: paradigm and anticipation - EPMA position paper 2016. EPMA J. 2016;7(1):23. https://doi.org/10.1186/s13167-016-0072-4.
- World Medical A. World Medical Association Declaration of Helsinki: ethical principles for medical research involving human subjects. JAMA. 2013;310(20):2191–4. https://doi.org/10.1001/jama.2013.281053.
- Reynolds JD and Olitsky SE, pediatric retina. 1st ed. Heidelberg: Springer-Verlag Berlin. 2010. https://doi.org/10.1007/978-3-642-12041-11.
- He K, Zhang X, Ren S, Sun J. Deep residual learning for image recognition. in Proceedings of the IEEE conference on computer vision and pattern recognition. 2016;770–778. https://doi.org/10.48550/arXiv.1512.03385.
- Tu Z, Talebi H, Zhang H, Yang F, Peyman Milanfar, Alan Bovik, et al. Maxvit: Multi-axis vision transformer. arXiv preprint arXiv. 2022: p. 01697. https://doi.org/10.48550/arXiv.2204.01697.
- Chen C, Rameswar P, Fan Q. Regionvit: Regional-to-local attention for vision transformers. arXiv preprint arXiv. 2022. https://doi.org/10.48550/arXiv.2106.02689.
- Lee SH, Lee S, Song BC. Vision transformer for small-size datasets. arXiv preprint arXiv. 2021. https://doi.org/10.48550/arXiv.2112.13492.
- Yu S, Ma K, Bi Q, Bian C. Mil-vt: Multiple instance learning enhanced vision transformer for fundus image classification[C]//Medical Image Computing and Computer Assisted Intervention-MICCAI 2021: 24th International Conference, Strasbourg, France, September 27-October 1, 2021, Proceedings, Part VIII 24. Springer International Publishing. 2021: p. 45-54. https://doi.org/10.1007/978-3-030-87237-3_5.
- Sun R, Li Y, Zhang T, Mao Z, Wu F. Lesion-aware transformers for diabetic retinopathy grading[C]. In Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. 2021: p. 10938–10947. https://doi.org/10.1109/CVPR46437.2021.01079.
- Playout C, Duval R, Boucher MC, Cheriet F. Focused Attention in Transformers for interpretable classification of retinal images. Med Image Anal. 2022;82:102608. https://doi.org/10.1016/j.media.2022.102608.
- Wong TY, Bressler NM. Artificial Intelligence With Deep Learning Technology Looks Into Diabetic Retinopathy Screening. JAMA. 2016;316(22):2366–7. https://doi.org/10.1001/jama.2016.17563.
- Ng WY, Zhang S, Wang Z, Ong CJT, Gunasekeran DV, Lim GYS, Ting DSW. Updates in deep learning research in ophthalmology. Clin Sci. 2021;135(20):2357–76.
- Huang YPBH, Kang EYC, Chen KJ, Hwang YS, Lai CC, Wu WC. Automated detection of early-stage ROP using a deep convolutional neural network. Br J Ophthalmol. 2021;105(8):1099–103.
- Durai C, Jebaseeli TJ, Alelyani S, Mubharakali A. Early Prediction and Diagnosis of Retinoblastoma Using Deep Learning Techniques. arXiv preprint arXiv. 2021. https://doi.org/10.48550/arXiv.2103.07622.
- Stevenson CH, Hong SC, Ogbuehi KC. Development of an artificial intelligence system to classify pathology and clinical features on retinal fundus images. Clin Exp Ophthalmol. 2019;47(4):484–9. https://doi.org/10.1111/ceo.13433.
- Son J, Shin JY, Kim HD, Jung KH, Park KH, Park SJ. Development and Validation of Deep Learning Models for Screening Multiple Abnormal Findings in Retinal Fundus Images. Ophthalmology. 2020;127(1):85–94. https://doi.org/10.1016/j.ophtha.2019.05.029.
- Lin D, Xiong J, Liu C, Zhao L, Li Z, Yu S, et al. Application of Comprehensive Artificial intelligence Retinal Expert (CARE) system: a national real-world evidence study. Lancet Digit Health. 2021;3(8):e486–95. https://doi.org/10.1016/S2589-7500(21)00086-8.
- Cen L-P, Ji J, Lin J-W, Ju S-T, Lin H-J, Li T-P, et al. Automatic detection of 39 fundus diseases and conditions in retinal photographs using deep neural networks. Nat Commun. 2021;12(1). https://doi.org/10.1038/s41467-021-25138-w.
- Ju L, Yu Z, Wang L, Zhao X, Wang X, Bonnington P, et al. Hierarchical Knowledge Guided Learning for Real-world Retinal Disease Recognition. IEEE Trans Med Imaging. 2023;PP. https://doi.org/10.1109/TMI.2023.3302473.
- Gu C, Wang Y, Jiang Y, Xu F, Wang S, Liu R, et al. Application of artificial intelligence system for screening multiple fundus diseases in Chinese primary healthcare settings: a real-world, multicentre and cross-sectional study of 4795 cases. Br J Ophthalmol. 2023. https://doi.org/10.1136/bjo-2022-322940.
- Zhu H, Cheng C, Yin H, Li X, Zuo P, Ding J, et al. Automatic multilabel electrocardiogram diagnosis of heart rhythm or conduction abnormalities with deep learning: a cohort study. Lancet Digit Health. 2020;2(7):e348–57. https://doi.org/10.1016/S2589-7500(20)30107-2.
- Golubnitschaja O, Topolcan O, Kucera R, Costigliola V, Epma. 10th Anniversary of the European Association for Predictive, Preventive and Personalised (3P) Medicine - EPMA World Congress Supplement 2020. EPMA J. 2020;11(Suppl 1):1–133. https://doi.org/10.1007/s13167-020-00206-1.
版权说明:
本文由 youcans@xidian 对论文 Automated detection of nine infantile fundus diseases and conditions in retinal images using a deep learning system 进行摘编和翻译。该论文版权属于原文期刊和作者,本译文只供研究学习使用。
youcans@xidian 作品,转载必须标注原文链接:
【医学影像 AI】使用深度学习系统检测 9 种婴儿眼底疾病和病症(https://youcans.blog.csdn.net/article/details/146136764)
Crated:2025-03
更多推荐
 已为社区贡献14条内容
已为社区贡献14条内容









所有评论(0)